纳米化学表征技术揭示单分子层表面功能化中的涂层缺陷
Get the pdf download to your inbox:
背景
表面修饰技术通过调控分子相互作用已成为一种重要手段。研究人员通过添加特定分子可结合的功能基团,能够精确控制抗体、酶、DNA、细菌及病毒与表面的相互作用[1]。该技术通常称为表面修饰或功能化,已广泛应用于生物技术、组织工程、生物传感器及半导体行业。其核心前提是功能基团需形成关键的单分子层以确保实验成功,因为表面的化学状态最终决定其与其他分子的相互作用。因此,功能化失败或污染物存在会引入干扰变量,导致异常实验结果难以排查。分析技术对于排除基底表面修饰问题所致的后续流程异常具有重要价值。
单分子层分析技术
尽管掌握修饰表面状态至关重要,但即使在先进显微与光谱技术支持下,纳米尺度表征仍存在挑战。理想技术需满足四大特性:非破坏性、常压环境操作、检测单分子层间隙的高空间分辨率,以及单分子层灵敏度。当前常用材料表征技术如飞行时间二次离子质谱(ToF-SIMS)与X射线光电子能谱(XPS)均存在局限:ToF-SIMS具破坏性,XPS虽具表面敏感性却需繁琐真空环境。遗憾的是,两者空间分辨率均存在不足。水接触角测量作为常用手段虽可便捷显示成功迹象,但因无法识别具体表面分子,可能导致成功假象。
此刻正是红外光诱导力显微镜(IR-PiFM)展现优势之时。凭借亚5纳米空间分辨率与亚单分子层灵敏度,IR-PiFM可精确测量有机/无机表面的红外吸收,在原始表面与功能化表面表征中均表现卓越[2]。后续两个表面修饰案例进一步验证其有效性:IR-PiFM不仅成功确认单分子层存在,更揭示了潜在表面相互作用机制。
案例研究一:氨基硅烷玻璃表面功能化
Get the pdf download to your inbox:
研究目标:
某研究团队采用氨基硅烷对玻璃基底进行功能化处理后,委托Molecular Vista公司检测其涂层质量。该材料在业界应用广泛,我们此前亦曾处理过类似商用基底的检测案例。使用PiFM技术对客户制备的样品进行了功能化前、后的对比分析。
结果:
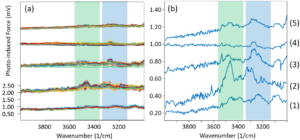
首先获取裸玻璃基底的2 µm × 1 µm形貌图(图1A),基于玻璃在1070 cm⁻¹的特征化学谱峰生成PiFM化学成像(图1B),直观呈现表面化学成分分布。随后在指定点位采集了六组PiF-IR光谱(对应图1A,B标注位点)。这些反映各点位分子物种红外特征的光谱,为揭示表面真实状态提供了重要信息。值得注意的是,所有光谱表现出高度重复性(图1C)。

形貌图像提示存在长程空间起伏的高度变化,但由于表面化学性质均一,这些特征未在PiFM图像中显现。PiFM图像中黄色虚线框区域的信号微弱区(PiF信号略低),很可能源于探针与玻璃基底间稍厚的污染物夹层。约2900 cm⁻¹处的小峰(对应2941 cm⁻¹与2869 cm⁻¹的C-H伸缩振动)及约1400 cm⁻¹处峰位(对应C-H弯曲振动)表明整个表面被有机污染物超薄层覆盖。由此可见,PiFM技术揭示了”裸”材料表观简单性下隐藏的复杂性。
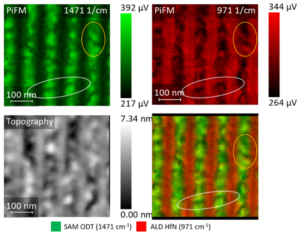
随后对氨基硅烷功能化玻璃基底进行分析,与首组裸玻璃样本对比。图2A展示2 µm × 2 µm形貌成像(第一步);基于1060 cm⁻¹(玻璃)、1623 cm⁻¹与1678 cm⁻¹三组PiFM图像的复合数据,生成综合PiFM图像(图2B)。
为完善分析,将图1C的六组光谱取平均值后作为虚线光谱显示于图2C用于对比,同时在氨基硅烷涂层玻璃基底图像标注点采集了六组PiF-IR光谱。形貌与PiFM图像的结合分析表明:氨基硅烷分子已聚集成簇而非形成单分子层组装体。

图2A形貌显示表面存在不同尺寸的聚集体。为解析玻璃基底及岛状结构的化学成分,采集了六组光谱:两组光谱(深蓝与绿色)采集自远离聚集体的裸基底区域,显示约1060 cm⁻¹处的主玻璃特征峰,表明大部分表面为纯净玻璃,同时可见约2900 cm⁻¹处先前识别的污染物峰(C-H伸缩振动)。
其余四组光谱(分别对应图2B,C中粉红、红色、金色及紫色标注的聚集体)显示以下特征峰:约1623 cm⁻¹(NH₂剪切振动)、约1678 cm⁻¹(未知峰)及813 cm⁻¹(NH₂摇摆振动)。三个较高聚集体还呈现3500 cm⁻¹处的宽峰(NH伸缩振动)。与最矮的粉红色聚集体相比,这三个聚集体的1623 cm⁻¹峰强度显著更高。结果总结参见表1。
| 位置 | 光谱颜色 | 特征峰位 |
| 裸基底 | 深蓝, 绿色 | 1060 cm−1 (玻璃), 2900 cm−1 (污染物) |
| 低矮聚集体 | 粉色 | 1623 cm−1 (NH2 剪切振动), 1678 cm−1 (未知), 813 cm−1 (NH2 摇摆振动) |
| 高密度聚集体(红色, 金色, 紫色) | Red, Gold, Purple | 1623 cm−1 (NH2 剪切振动), 1678 cm−1 (未知), 813 cm−1 (NH2 摇摆振动), 3500 cm−1 (NH 伸缩振动) |

图3A形貌图像显示存在多个高度约0.7 nm至1.4 nm的小型团簇(紫色光谱采集位点的聚集体除外)。这些高度值能更准确反映团簇尺寸,因为探针针尖曲率会导致横向尺寸扩张。值得注意的是,凭借PiFM的高空间分辨率,即使在这些微小团簇上也能清晰观测胺基特征峰:红色光谱采集自仅约0.7 nm厚的团簇,其很可能为单层或双层结构。
值得关注的是,在这些较薄团簇上,光谱与图像均显示1679 cm⁻¹处峰强高于1628 cm⁻¹。这可能是由于分子形成了更有序的二维层状排列。该发现具有重要价值,因为PiFM技术基于探针取向特性对平面外振动更加敏感,从而为分子排列方式提供关键线索(关于晶体样品取向依赖性PiFM信号的详细说明参见应用指南)。
远离团簇区域的浅蓝色光谱(图2C)仅显示玻璃峰与污染物峰,无氨基硅烷特征峰。这证实大部分表面未被氨基硅烷覆盖。虽然当前状态可能满足某些应用需求,但形成更均匀的单分子层将显著提升最终产品的良率与可靠性。使用PiFM等技术识别单分子层中的空隙,有助于排查后续工艺中的异常问题。
案例研究二:硅基底表面功能化
研究目标:
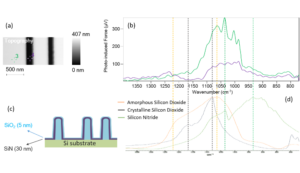
本研究通过第二个案例探究环氧终止的甲氧基硅烷在带自然氧化层的硅基底上的应用效果, specifically采用PiFM技术评估该表面处理的均匀性。

结果:
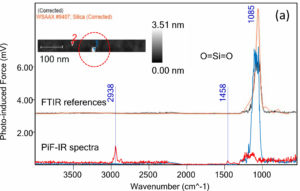
图4A的5 µm × 5 µm形貌图显示与案例一相似的分子聚集体。图4B中绿色标记点对应图4C绿色光谱采集位点。与基底光谱(紫色)相比,该光谱在1080 cm⁻¹与940 cm⁻¹处出现额外峰位,我们分别将其归属为Si-O-Si振动与Si-OH振动[3]。对应波数的PiFM图像显著凸显这些聚集体。此外可见更微弱的聚集体(白圈标注),其高度约0.5 nm(未显示),很可能为单层或双层聚集体。
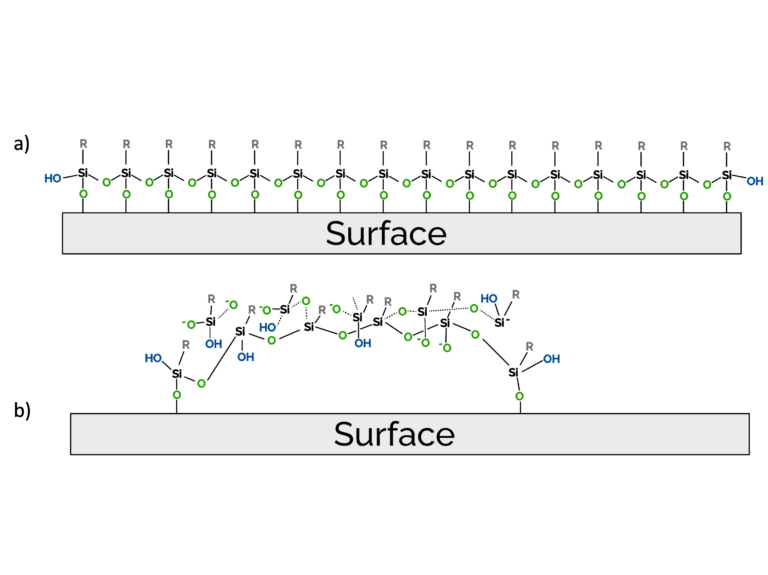
我们的观测与Naik等人提出的模型部分吻合:硅烷并非形成均匀单分子层(图5A),而是聚集成类似雪墩的结构(图5B)。这些团簇可能通过Si-O-Si键连接,仅少数硅烷分子实际附着于基底表面。
虽然Naik等人将团簇形成主要归因于烷基链范德华半径与Si-O-Si键距不匹配导致的分子内应变,但我们认为-OH键密度与氧化物表面清洁度同样起重要作用。

总结
我们的研究表明:两种表面处理均未形成预期的均匀单分子层,而是在基底上组装成团簇结构。这导致表面存在显著裸露区域,可能影响依赖这些功能基团相互作用的最终产品性能。需进一步研究调控团簇形成的关键因素,探索实现更均匀、更可靠表面改性的策略,并可通过PiFM技术进行验证分析。
参考文献
- Mukesh Digambar Sonawane and Satish Balasaheb Nimse, “Surface Modification Chemistries of Materials Used in Diagnostic Platforms with Biomolecules”, Journal of Chemistry, Volume 2016, Article ID 9241378
- Nowak, D., Morrison, W., Wickramasinghe, HK., Jahng, J., Potma, E., Wan, L., Ruiz, R., Albrecht, TR., Schmidt, K., Frommer, J., Sanders, DP., Park, S., “Nanoscale chemical imaging by photoinduced force microscopy”, Sci. Adv., 2:e1501571 (2016).
- Ahmed A. Issa 1 and Adriaan S. Luyt, “Kinetics of Alkoxysilanes and Organoalkoxysilanes Polymerization: A Review”, Polymers 2019, 11, 537; doi:10.3390/polym11030537
- Naik, V., Crobu, M., Venkataraman, V., Spencer, N., “Multiple Transmission-Reflection IR Spectroscopy Shows That Surface Hydroxyls Play Only a Minor Role in Alkylsilane Monolayer Formation on Silica”, J. Phys. Chem. Lett. 2013, 4, 2745-2751; dx.doi.org/10.1021/jz401440d
Interested in a niche application?
Ask us, we may have already studied it.